FOR-COVID
Bayerischer Forschungsverbund zur Eindämmung, Behandlung und Erforschung der Erkrankung mit dem neuartigen Coronavirus COVID-19 (FOR-COVID)

Identifizierung der molekularen Grundlagen einer SARS-CoV-2 Infektion mit Hilfe einer neuen RNA Sequenzierungsmethode und künstlicher Intelligenz
Arbeitsfeld:
Erforschung der Virus-Zell Interaktion: Pathogeneseforschung und Erforschung neuer Therapieansätze
Projektleiter
Prof. Dr. Lars Dölken
Dr. Florian Erhard
Dr. Antoine-Emmanuel Saliba
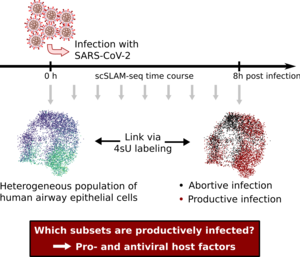
Auf Ebene einzelner Zellen ist das Ergebnis einer SARS-CoV-2 Infektion sehr heterogen: In einem Teil der Zellen werden innerhalb weniger Stunden enorme Mengen viraler RNAs exprimiert, wohingegen andere Zellen die Infektion kontrollieren können. Die Ursachen und Mechanismen hinter diesem dichotomen Fortschreiten der Infektion sind unbekannt. Ziel unseres Projektes ist es mit Hilfe einer von uns neu-entwickelten Technologie zur zeitlich aufgelösten Sequenzierung der RNA einzelner Zellen molekulare Faktoren zu finden, die den Infektionsverlauf maßgeblich beeinflussen. Wir werden dazu sowohl etablierte Zell-Linien als auch primäre, ex vivo infizierte Spenderzellen über mehrere Zeitpunkte der Infektion untersuchen, und SARS-Cov-2 mit zwei weiteren relevanten humanpathogenen respiratorischen Viren (Influenza und RSV) vergleichen. Zur Analyse und Integration der gewonnenen Daten werden neu entwickelte KI-Methoden zum Einsatz kommen. Wir werden dabei eng mit der AG Prof. Pichlmair und der AG Prof Vogel / Prof.Munschauer zusammenarbeiten, um speziell Proteine, die mit dem Virusgenom interagieren und Interferon-regulierte Gene als Kandidaten zu untersuchen. Unser Projekt wird die molekularen Grundlagen der Wechselwirkung zwischen Virus und Wirtszelle erforschen und potenzielle Faktoren identifizieren, die produktive Infektion verhindern können.
Projektpartner:
- Universität Regensburg
- Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) Würzburg
- Julius-Maximilians-Universität Würzburg



